
Firstname name
Function
I'm a paragraph. I'm connected to your collection through a dataset. To update me, go to the Data Manager. The Data Manager is where you store data to use in your site pages, or collect data from site visitors when they submit a form.
This collection in the Data Manager is already set up with some fields and content. To customize it with your own content, you can import a CSV file or simply edit the placeholder text. You can also add more fields which you can connect to other page elements so the content displays on your published site. Remember to sync the collection so your content is live! You can add as many new collections as you need to store or collect data.

Firstname name
Function
I'm a paragraph. I'm connected to your collection through a dataset. To update me, go to the Data Manager. The Data Manager is where you store data to use in your site pages, or collect data from site visitors when they submit a form.
This collection in the Data Manager is already set up with some fields and content. To customize it with your own content, you can import a CSV file or simply edit the placeholder text. You can also add more fields which you can connect to other page elements so the content displays on your published site. Remember to sync the collection so your content is live! You can add as many new collections as you need to store or collect data.

Firstname name
Function
I'm a paragraph. I'm connected to your collection through a dataset. To update me, go to the Data Manager. The Data Manager is where you store data to use in your site pages, or collect data from site visitors when they submit a form.
This collection in the Data Manager is already set up with some fields and content. To customize it with your own content, you can import a CSV file or simply edit the placeholder text. You can also add more fields which you can connect to other page elements so the content displays on your published site. Remember to sync the collection so your content is live! You can add as many new collections as you need to store or collect data.

Firstname name
Function
I'm a paragraph. I'm connected to your collection through a dataset. To update me, go to the Data Manager. The Data Manager is where you store data to use in your site pages, or collect data from site visitors when they submit a form.
This collection in the Data Manager is already set up with some fields and content. To customize it with your own content, you can import a CSV file or simply edit the placeholder text. You can also add more fields which you can connect to other page elements so the content displays on your published site. Remember to sync the collection so your content is live! You can add as many new collections as you need to store or collect data.

Firstname name
Function
I'm a paragraph. I'm connected to your collection through a dataset. To update me, go to the Data Manager. The Data Manager is where you store data to use in your site pages, or collect data from site visitors when they submit a form.
This collection in the Data Manager is already set up with some fields and content. To customize it with your own content, you can import a CSV file or simply edit the placeholder text. You can also add more fields which you can connect to other page elements so the content displays on your published site. Remember to sync the collection so your content is live! You can add as many new collections as you need to store or collect data.

Firstname name
Function
I'm a paragraph. I'm connected to your collection through a dataset. To update me, go to the Data Manager. The Data Manager is where you store data to use in your site pages, or collect data from site visitors when they submit a form.
This collection in the Data Manager is already set up with some fields and content. To customize it with your own content, you can import a CSV file or simply edit the placeholder text. You can also add more fields which you can connect to other page elements so the content displays on your published site. Remember to sync the collection so your content is live! You can add as many new collections as you need to store or collect data.

Firstname name
Function
I'm a paragraph. I'm connected to your collection through a dataset. To update me, go to the Data Manager. The Data Manager is where you store data to use in your site pages, or collect data from site visitors when they submit a form.
This collection in the Data Manager is already set up with some fields and content. To customize it with your own content, you can import a CSV file or simply edit the placeholder text. You can also add more fields which you can connect to other page elements so the content displays on your published site. Remember to sync the collection so your content is live! You can add as many new collections as you need to store or collect data.
The ASTR team aims to produce knowledge in genetics to adapt sunflower to the challenges of agro-ecological transition.
Sunflower is the 4th most important oilseed in the world and the 2nd most important in France. Its low water and input requirements as well as its short cycle give it a very interesting ecological profile. It is an essential component of future agricultural systems (Debaeke et al. 2021), and its production has increased by 44% in the last 10 years (source USDA).
Mainly cultivated in environments subject to water stress, we are interested in
- the genetic control of yield plasticity in relation to water stress, cold following early sowing to avoid drought
- molecular mechanisms involved in responses to abiotic stresses in innovative cropping systems
- the impact of climate change on the attractiveness of sunflower for pollinators
To carry out our work, we are developing resources at three levels:
- at the genetic level with the CRB Tournesol
- at the genomic level with the ICSG
- at the phenomenal level, the high-throughput phenotyping platform Heliaphen which is part of the local infrastructure Phenotoul
MEMBERS
EU RIA HELEX
HelEx est un projet de recherche et d’innovation de 4 ans soutenu par le programme Horizon Europe de l’UE.
Le projet de 5,5 millions d’euros vise à identifier des stratégies moléculaires et génétiques permettant d’adapter la culture du tournesol au changement climatique tout en améliorant son empreinte environnementale.
L’objectif principal est de créer de nouvelles variétés de tournesol plus résistantes à la sécheresse et aux conditions climatiques extrêmes, en utilisant des variétés d’Helianthus Extremophilus.
HelEx : améliorer la résilience du tournesol face aux défis climatiques
Les stress abiotiques, en particulier la sécheresse et la chaleur, ont un impact significatif sur la physiologie et le développement des plantes et, par conséquent, sur le rendement des cultures.
En Europe, la sécheresse a un effet prononcé sur l’agriculture, notamment pour les producteurs de tournesol. Les espèces végétales sauvages, en particulier les 49 espèces d’Helianthus originaires d’Amérique du Nord, ont évolué pour résister à de tels stress.
Ces espèces ont développé des stratégies de résilience pour prospérer dans des environnements extrêmes, tels que les déserts et les zones de haute altitude. Elles restent très attractives pour les pollinisateurs, ce qui est crucial puisqu’elles sont auto-incompatibles. L’attrait du tournesol pour les pollinisateurs est conditionné par des signaux visuels et olfactifs, ainsi que par des récompenses comme le nectar et le pollen, susceptibles d’être affectées par les conditions environnementales.
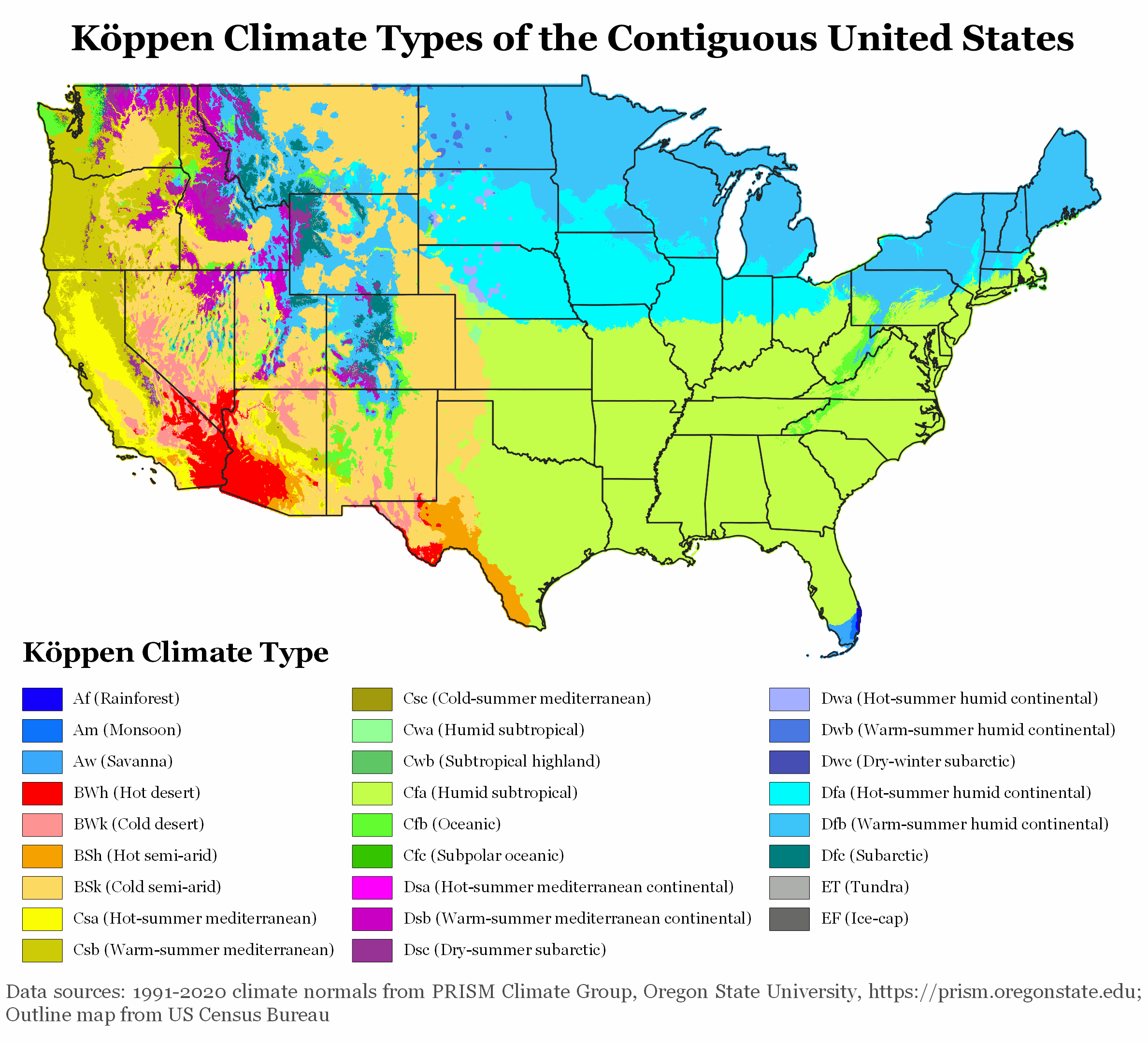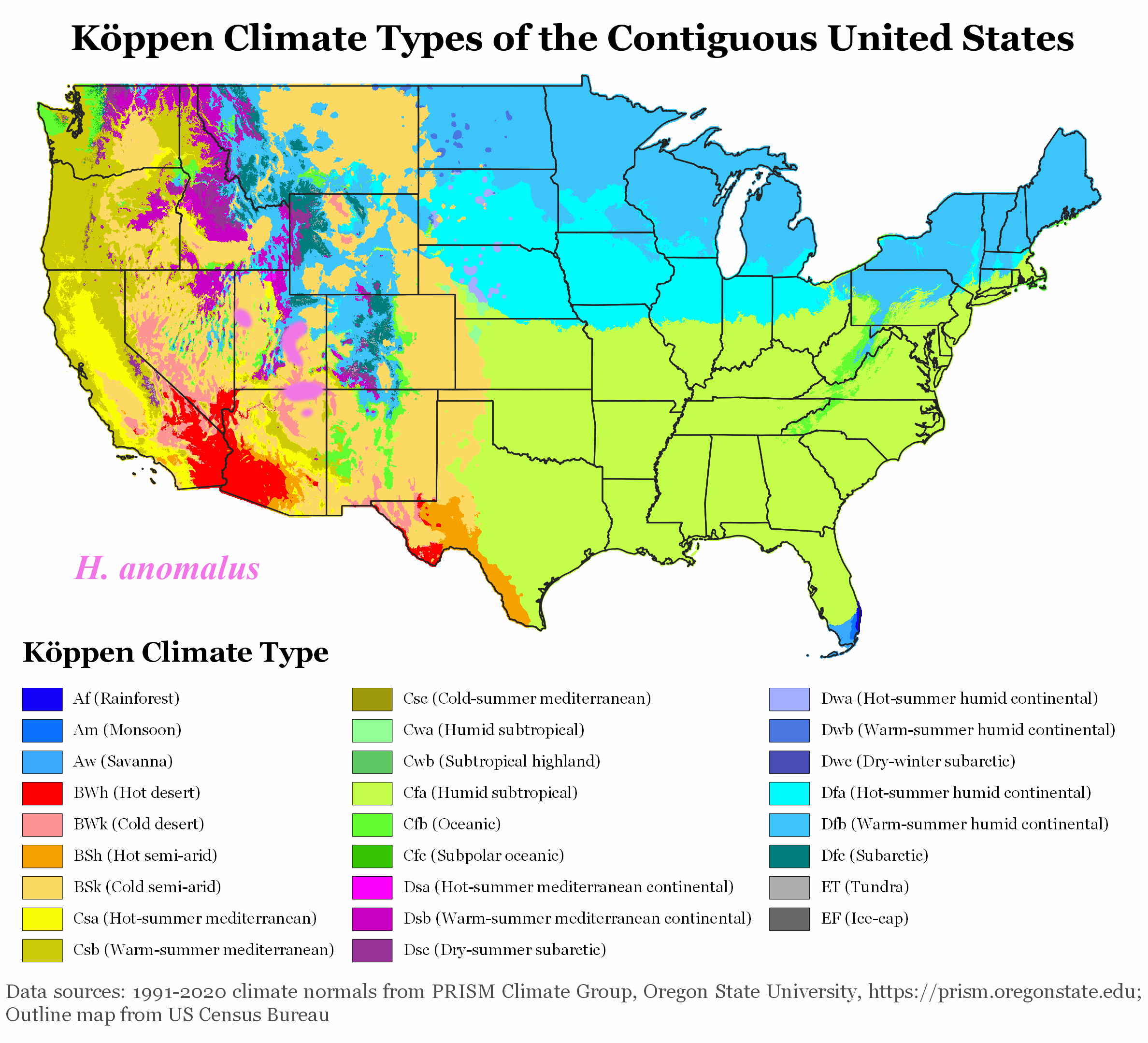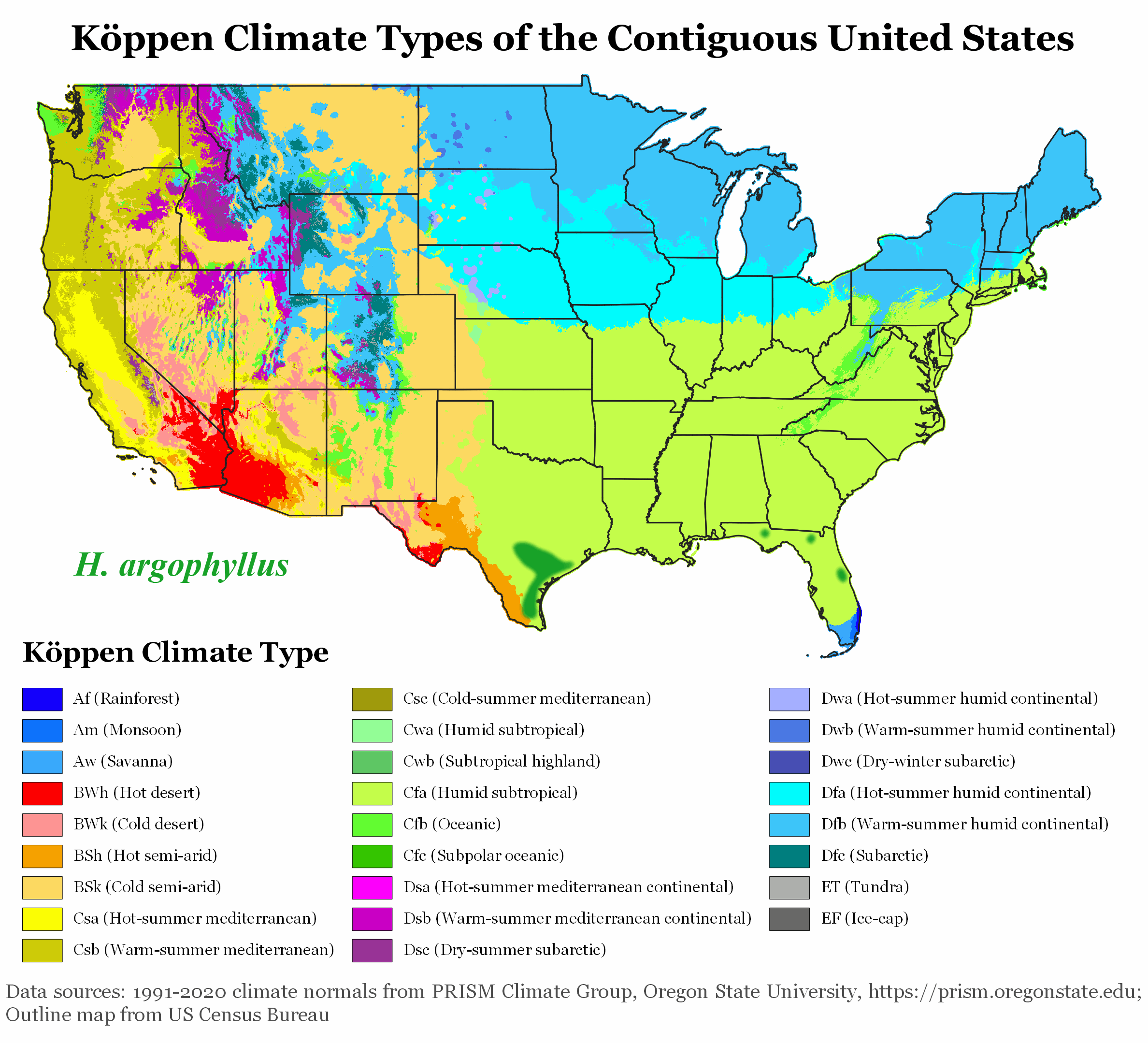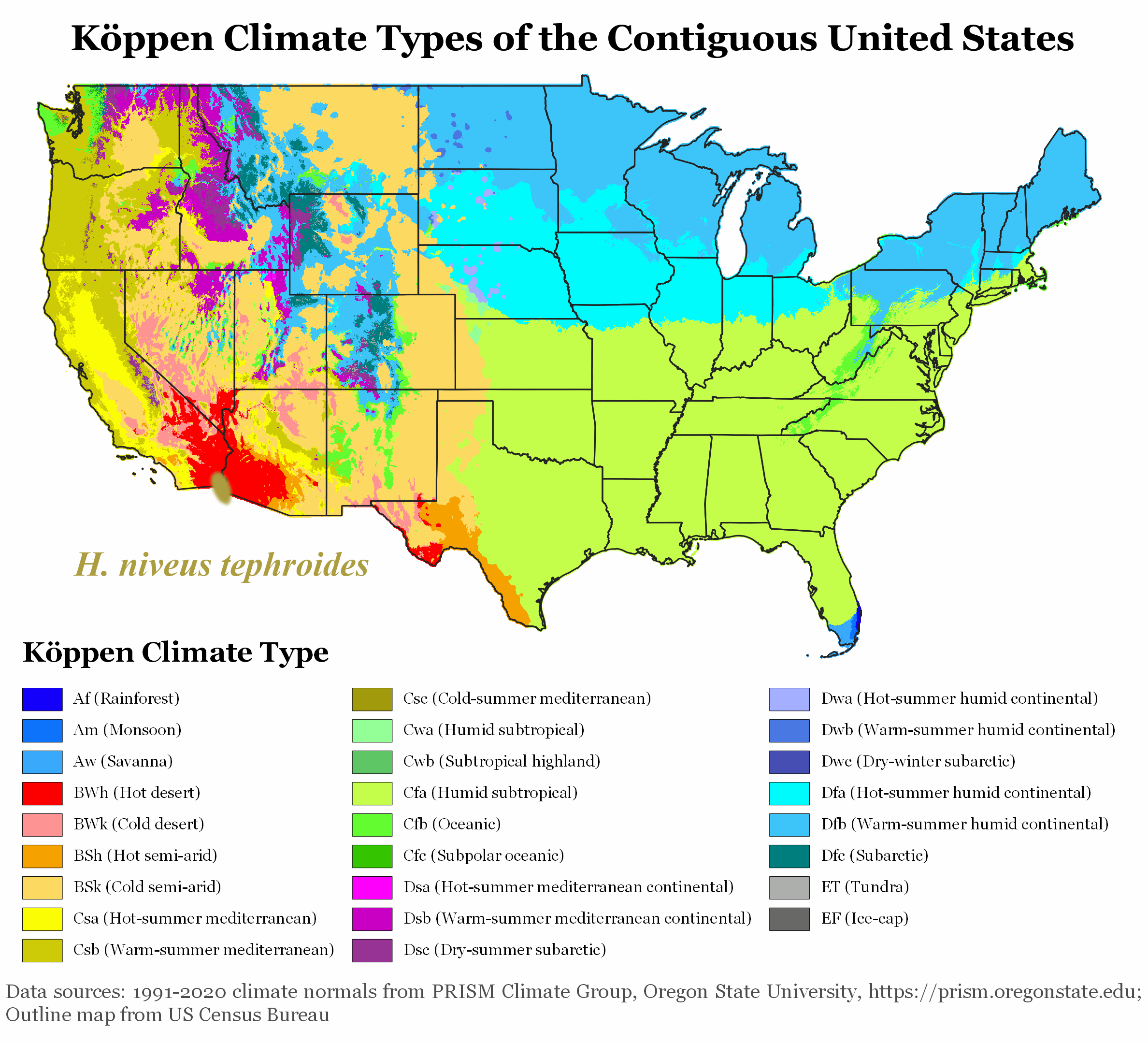

Améliorer la résilience du tournesol grâce à la génétique sauvage et aux technologies de pointe
HelEx vise à exploiter les mécanismes génétiques et moléculaires développés par les espèces sauvages d’Helianthus pour s’adapter aux climats extrêmes.
L’objectif est d’intégrer ces mécanismes dans les tournesols cultivés afin de maintenir la qualité des semences et les ressources en pollinisateurs. Plusieurs projets se sont concentrés sur la tolérance du tournesol à la sécheresse et à d’autres stress abiotiques, principalement au niveau moléculaire.
HelEx cherche à aller au-delà des connaissances existantes en développant des méthodes de phénotypage innovantes utilisant l’IA, en produisant des réseaux multi-omiques et en identifiant de nouveaux haplotypes à partir d’espèces sauvages d’Helianthus.
Le projet vise également à améliorer les programmes de sélection du tournesol, en relevant des défis tels que l’étroite base génétique du tournesol cultivé et la nature complexe des caractéristiques souhaitées.
Les 18 partenaires de HelEx
Les partenaires de HelEx associent les instituts de recherche académiques en pointe sur le tournesol et les entreprises semencières leader mondiales pour les semences de tournesol de 7 pays européens France, Allemagne, Pays-Bas, Serbie, Autriche, Roumanie, Pologne, et du Canada et des Etats-Unis.

PIA SUNRISE
Le projet SUNRISE, SUNflower Resources to Improve yield Stability in a changing Environment, a pour objectif de développer les connaissances, les ressources et les outils sur le tournesol pour permettre d’adapter cette culture aux challenges du changement climatique notamment en maintenant sa productivité dans des conditions de sécheresse.
Le projet articule plusieurs disciplines : la génétique, la génomique, la physiologie, l’agronomie et les sciences sociales. Il regroupe les acteurs de la recherche (9 laboratoires INRAE et universitaires), l’institut technique sur les oléoprotéagineux et 6 entreprises en biotechnologie et semencières dans le but non pas de travailler pour mais avec les acteurs de terrain et d’assurer un transfert plus rapide et efficace des connaissances, méthodes et ressources produites.
Le projet a un fort ancrage dans le sud-ouest et à Toulouse en particulier ce qui a renforcé la visibilité de Toulouse comme capitale mondiale de la recherche et de la sélection sur le tournesol.
SUNRISE, a constitué un projet central de l’équipe ASTR entre 2012 et 2020 et se poursuit à travers l’analyse des données produites et l’exploitation des pistes de recherches ouvertes par ce projet d’envergure.

Sunrise en chiffres :
Budget
Total : 21M€, Subvention : 7M€ (INRAE 4.3M€), Autofonancement public : 7M€ (INRAE 4.7M€) et autofinancement privé 7M€
Financement : Agence Nationale de la Recherche ANR-11-BTBR-005
Dates : Septembre 2012 - Décembre 2020, 8 ans et 4 mois
Coordination : Patrick Vincourt (2012-2013) et Nicolas Langlade (2014-2020)
16 parternaires :
-
9 laboratoires publics INRAE LIPME, INRAE AGIR, INRAE MIAT, INRAE CNRGV, INRAE EPGV, Sorbonne Université, INRAE GQE, Université Toulouse 1 LEREPS, INRAE BFP
-
1 institut technique : Terres Inovia
-
1 entreprise en biotechnologie : Innolea
-
5 semenciers : Caussade Semences puis Lidea, Mas Seeds, RAGT, , Soltis, Syngenta
Personnel
Personnel public : 86 personnes dont 32 chercheurs, 3 thèses, 10 post-docs
Personnel privé : environ 100 scientifiques
Données produites
~100 expérimentations : 45 expérimentations en champ en France, Roumanie Chili et Argentine, ~60 sur plateformes de phénotypage Phenotoul
~6700 génotypes de tournesol créés ou utilisés
7020 plantes phénotypées sur Heliaphen
>2000M de données de génotypage SNP par puces et reséquençage
~1M de données agronomiques disponibles dans la base de données SUNRISE
5M de mesures de quantité de transcrits
1.2M de niveaux de méthylation
850k mesures de quantité de métabolites
172k mesures de quantité de protéines
Résultats marquants
1 - Séquençage du premier génome de référence du tournesol en 2017 (Badouin et al ., ,2017 Nature). Issue d’une collaboration étroite du LIPME, CNRGV, GeT pour acquérir un séquenceur de nouvelle génértation et casser le verrou de l’assemblage des grands génomes complexes de plantes. Deux autres génomes du tournesol ont été séquencés ensuite dans SUNRISE. La méthodologie développée est devenue un standard en génomique a été largement réutilisée pour les séquençages de dizaines de plantes, animaux et microorganismes (rosier, Medicago, vigne, orobanche, homme, macaque, abeille, etc…).
2 – Identification du contrôle génétique de la tolérance à la sécheresse (Mangin et al.,, 2017 et Gosseau et al., 2019 et en cours de valorisation). Une approche combinée de génétique quantitative et de modélisation agronomique développée au LIPME et à AGIR et grâce aux essais des partenaires privés a permis d’identifier les types de tournesol adaptés à différents climats en Europe et aux futurs scénarios climatiques. L’intégration de ces résultats avec la modéliasation des régulations génétiques à MIAT a permis de mettre en évidence les réseaux de gènes impliqués dans la tolérance à la sécheresse qui ont été travaillés par la sélection moderne qu’il faut continuer à améliorer pour les tournersol du futur.
3- Développement d’outils de phénotypage haut-débit (Gosseau et al., 2019). Une étroite collaboration entre l’UEGCA, le LIPME et le PIA PHENOME a été permise par SUNRISE et a permis le développement des premiers outils de phénotypage haut-débit sur le tournesol appliqués sur le plus grand essai de tournesol mené à l’INRAE en 2017 (1800 parcelles sur 3ha du domaine de Langlade caractérisées en drone). Cela a également abouti à la création de l’infrastructure collective Phenotoul qui concentre sur un même site, de façon unique au monde, un continuum de la serre, à la plateforme semi-contrôlée jusqu’au champ avec TPMP, Heliaphen et Agrophen.
4 –Eco-innovations pour la filière semencière et intégration dans le système d’évaluation des variétés (Galliano et al. 2017) SUNRISE a permis de conceptualiser les éco-innovations dans le domaine de l’amélioration variétale et d’étudier les verrous de l’acceptation de ces innovations par les agriculteurs au niveau français et européen par un travail collaboratif entre AGIR et le LEREPS et en s’appuyant sur les réseaux internationaux des semenciers du projet.
5 – Développement de matériel génétique incorporant la diversité génétique sauvage. Les ressources génétiques produites dans le cadre de SUNRISE sont uniques : le Centre de Ressources Biologiques du Tournesol basé au LIPME a développé avec les partenaires semenciers 2437 lignées recombinantes, 448 lignées d’introgression avec du tournesol sauvage. SUNRISE a permis leur caractérisation moléculaire : ~3500 lignées génotypées à haute densité, ce qui prépare l’exploitation des ressources génétiques dans de futurs projets.
Abiotic stress tolerance (Coordinator: Nicolas Langlade)
Identification of molecular and functional polymorphisms, in cultivated and wild sunflowers, in candidate genes involved in tolerance to abiotic stress and in development. Gene selection was performed mainly in the model species Arabidopsis thaliana.
Genetic analysis of (1) oil content under water stress, and (2) physiological and developmental responses to moderate and severe water stress in the field and under controlled conditions using association genetics and QTL mapping approaches . This work aims to integrate genetic modeling into the Sunflo agronomic model (Casadebaig et al. 2011)
A systems biology approach to transcriptomic regulatory pathways of hormonal signals and abiotic stresses. By using either variations in hormones and duration, or the genetic variability of the response to hormones, we reconstruct gene regulatory networks in relation to limiting factors in sunflower cultivation such as the regulation of perspiration (mediated by ABA), leaf senescence or flowering time. These models are then compared to observations of responses to abiotic stresses and to natural variability and in the cultivated pool to identify how the environment interacts with gene networks at short (responses of an organism) and long (evolution and domestication) scales. .
Resistance to Orobanche cumana, the sunflower broomrape (Coordinator: Stéphane Muños)
Mapping and cloning of total and quantitative resistance to the most virulent races of Orobanche.
Physiological characterization of the interaction between sunflower and O. cumana
Measurement of gene expression during the early stages of the interaction
New statistical strategies for Association Genetics and Genomics selection (coordinator: Brigitte Mangin)
New models for Association genetics and construction of confidence regions of detected QTLs.
Improving the accuracy of predictions of the value of complex characters in plant species of agronomic interest.
Resource development and maintenance
Public
-
“Core collections”: http://www.heliagene.org/Web/public/core/Core_collections_list.html
-
Genetic populations developed by Felicity Vear (INRA Clermont-Ferrand) and Hervé Serieys (INRA Montpellier)
-
Bioinformatics resources (with strong support from the LIPM Bioinformatics team ):
-
HELIAGENE Portal (Carrere et al., 2008): an atlas of annotated sunflower genes based on publicly available data as of September 2007, assembled by Mike Barker (U. British Columbia, Vancouver) http://www.heliagene.org
-
"H + P" portal: TSEs annotated with sunflower seedlings infected by Plasmopara halstedii: http://www.heliagene.org/HP
-
Resources available under contract or in collaboration:
-
population of TILLING
-
Bioinformatics Resources (with strong support from the LIPM Bioinformatics team ):
-
Resequencing of the genome of INRA sunflower lines (including the INRA XRQ reference line)
-
Gene atlas (mRNA and sRNA transcriptome) of the INRA XRQ reference line
-
Database of SNP or indel polymorphisms developed from candidate genes and identified on cultivated and wild sunflowers
-
P.halstedii transcriptome (four races)
-
Collaborations
Sunflower genome sequencing project: University of British Columbia (Canada, L. Rieseberg): (Kane et al., 2010, 2011)
ACT: http://wwwagir.toulouse.inra.fr/agir/ group “Vasco” team: Ph.Debaeke
BIA: http://carlit.toulouse.inra.fr/wikiz/index.php/ Home group SaAB (Statistics and Algorithmics for Biology) B. Mangin and M. Vignes
URGV: S. Balzergues: http://www-urgv.versailles.inra.fr/
CNRGV: H.Berges, http://cnrgv.toulouse.inra.fr/en
Terres inovia: http://www.terresinovia.fr/ E.Mestries.
INRA: F. Delmotte (Bordeaux), P.Mestre (Colmar)
Financial ressources
SUNRISE: Project selected within the framework of "Investments for the Future" (wave 2011), bringing together 10 public research teams, CETIOM and 6 industrial partners involved in the genetic improvement of sunflowers. Project coordination by the LIPM team. Global funding: € 7 million, of which € 2.6 million for the LIPM. Duration: 7.4 years (2012-2019)
OLEOSOL (labeled by AGRIMIP Innovation http://www.agrimipinnovation.com/en/ ) funded by the Midi-Pyrénées Region, the FEDER and the FUI. Partnership with BIOGEMMA, SYNGENTA Seeds, SOLTIS, R2N: “Tools and resources towards sunflower genetic improvement for oil yield production. »€ 1,296k
AIP INRA Bioressources 2009 and 2010, INRA GAP Department, 2010: Contribution to sunflower genome sequencing. € 248K
[2009-2011]: PROMOSOL: Genetic analysis of tolerance to Phoma macdonaldii, 30K €
[2013-2014]: Sunflower * Orobanche interaction,
[2013-2015]: HELIADIV: sequence polymorphism analysis for at least 200 candidate genes involved in the response to abiotic and biotic stresses, in a large collection of lines (1000 accessions) and within various genetic pools, the whole of these genetic resources forming part of the collection of genetic resources managed by the team.
CETIOM: Phenotypic assessment of quantitative resistance to Downy Mildew Plasmopara halstedii. 17K €
scientific output
Adiredjo, AL, Casadebaig, P., Langlade, N., Lamaze, T., Grieu, P., 2018. Genetic Analysis of the Transpiration Control in Sunflower (Helianthus Annuus L) Subjected to Drought. VEGETOS: An International Journal of Plant Research 2018. https://doi.org/10.4172/2229-4473.1000368
Adiredjo, AL, Navaud, O., Muños, S., Langlade, NB, Lamaze, T., Grieu, P., 2014. Genetic Control of Water Use Efficiency and Leaf Carbon Isotope Discrimination in Sunflower (Helianthus annuus L.) Subjected to Two Drought Scenarios. PLoS ONE 9, e101218.
Andrianasolo, FN, Casadebaig, P., Langlade, N., Debaeke, P., Maury, P., 2016. Effects of plant growth stage and leaf aging on the response of transpiration and photosynthesis to water deficit in sunflower. Functional Plant Biology 43, 797–805.
Badouin, H., Gouzy, J., Grassa, CJ, Murat, F., Staton, SE, Cottret, L., Lelandais-Brière, C., Owens, GL, Carrère, S., Mayjonade, B., Legrand , L., Gill, N., Kane, NC, Bowers, JE, Hubner, S., Bellec, A., Bérard, A., Bergès, H., Blanchet, N., Boniface, M.-C., Brunel, D., Catrice, O., Chaidir, N., Claudel, C., Donnadieu, C., Faraut, T., Fievet, G., Helmstetter, N., King, M., Knapp, SJ, Lai , Z., Le Paslier, M.-C., Lippi, Y., Lorenzon, L., Mandel, JR, Marage, G., Marchand, G., Marquand, E., Bret-Mestries, E., Morien , E., Nambeesan, S., Nguyen, T., Pegot-Espagnet, P., Pouilly, N., Raftis, F., Sallet, E., Schiex, T., Thomas, J., Vandecasteele, C. , Varès, D., Vear, F., Vautrin, S., Crespi, M., Mangin, B., Burke, JM, Salse, J., Muños, S., Vincourt, P., Rieseberg, LH, Langlade , NB, 2017. The sunflower genome provides insights into oil metabolism, flowering and Asterid evolution. Nature 546, 148–152. https://doi.org/10.1038/nature22380
Balliau, T., Duruflé, H., Blanchet, N., Blein-Nicolas, M., Langlade, NB, Zivy, M., 2021. Proteomic data from leaves of twenty-four sunflower genotypes under water deficit. OCL 28, 12. https://doi.org/10.1051/ocl/2020074
Berton, T., Bernillon, S., Fernandez, O., Duruflé, H., Flandin, A., Cassan, C., Jacob, D., Langlade, NB, Gibon, Y., Moing, A., 2021 Leaf metabolomic data of eight sunflower lines and their sixteen hybrids under water deficit. OCL 28, 42. https://doi.org/10.1051/ocl/2021029
Blanchet, N., Casadebaig, P., Debaeke, P., Duruflé, H., Gody, L., Gosseau, F., Langlade, NB, Maury, P., 2018. Data describing the eco-physiological responses of twenty -four sunflower genotypes to water deficit. Data Brief 21, 1296–1301. https://doi.org/10.1016/j.dib.2018.10.045
Bonnafous, F., Fievet, G., Blanchet, N., Boniface, M.-C., Carrère, S., Gouzy, J., Legrand, L., Marage, G., Bret-Mestries, E., Munos, S., Pouilly, N., Vincourt, P., Langlade, N., Mangin, B., 2018. Comparison of GWAS models to identify non-additive genetic control of flowering time in sunflower hybrids. Theor. Appl. Broom. 131, 319–332. https://doi.org/10.1007/s00122-017-3003-4
Bordat, A., Marchand, G., Langlade, NB, Pouilly, N., Muños, S., Dechamp-Guillaume, G., Vincourt, P., Bret-Mestries, E., 2017. Different genetic architectures underlie crop responses to the same pathogen: the {Helianthus annuus * Phoma macdonaldii} interaction case for black stem disease and premature ripening. BMC Plant Biology 17, 167. https://doi.org/10.1186/s12870-017-1116-1
Debaeke, P., Casadebaig, P., Flenet, F., Langlade, N., 2017. Sunflower crop and climate change: vulnerability, adaptation, and mitigation potential from case-studies in Europe. OCL 24, D102. https://doi.org/10.1051/ocl/2016052
Debaeke, P., Casadebaig, P., Langlade, NB, 2021. New challenges for sunflower ideotyping in changing environments and more ecological cropping systems. OCL 28, 29. https://doi.org/10.1051/ocl/2021016
Fernandez, O., Urrutia, M., Bernillon, S., Giauffret, C., Tardieu, F., Le Gouis, J., Langlade, N., Charcosset, A., Moing, A., Gibon, Y. , 2016. Fortune telling: metabolic markers of plant performance. Metabolomics 12, 158.
Fernandez, O., Urrutia, M., Berton, T., Bernillon, S., Deborde, C., Jacob, D., Maucourt, M., Maury, P., Duruflé, H., Gibon, Y., Langlade, NB, Moing, A., 2019. Metabolomic characterization of sunflower leaf allows discriminating genotype groups or stress levels with a minimal set of metabolic markers. Metabolomics 15, 56. https://doi.org/10.1007/s11306-019-1515-4
Gascuel, Q., Bordat, A., Sallet, E., Pouilly, N., Carrere, S., Roux, F., Vincourt, P., Godiard, L., 2016a. Effector Polymorphisms of the Sunflower Downy Mildew Pathogen Plasmopara halstedii and Their Use to Identify Pathotypes from Field Isolates. PLoS ONE 11, e0148513. https://doi.org/10.1371/journal.pone.0148513
Gascuel, Q., Buendia, L., Pecrix, Y., Blanchet, N., Muños, S., Vear, F., Godiard, L., 2016b. RXLR and CRN effectors from the sunflower downy mildew pathogen Plasmopara halstedii induce hypersensitive-like responses in resistant sunflower lines. Forehead. Plant Sci. 7. https://doi.org/10.3389/fpls.2016.01887
Gody, L., Duruflé, H., Blanchet, N., Carré, C., Legrand, L., Mayjonade, B., Muños, S., Pomiès, L., Givry, S. de, Langlade, NB, Mangin, B., 2020a. Transcriptomic data of leaves from eight sunflower lines and their sixteen hybrids under water deficit. OCL 27, 48. https://doi.org/10.1051/ocl/2020044
Gosseau, F., Blanchet, N., Varès, D., Burger, P., Campergue, D., Colombet, C., Gody, L., Liévin, J.-F., Mangin, B., Tison, G., Vincourt, P., Casadebaig, P., Langlade, N., 2019. Heliaphen, an Outdoor High-Throughput Phenotyping Platform for Genetic Studies and Crop Modeling. Forehead. Plant Sci. 9. https://doi.org/10.3389/fpls.2018.01908
Hübner, S., Bercovich, N., Todesco, M., Mandel, JR, Odenheimer, J., Ziegler, E., Lee, JS, Baute, GJ, Owens, GL, Grassa, CJ, Ebert, DP, Ostevik , KL, Moyers, BT, Yakimowski, S., Masalia, RR, Gao, L., Ćalić, I., Bowers, JE, Kane, NC, Swanevelder, DZH, Kubach, T., Muños, S., Langlade, NB, Burke, JM, Rieseberg, LH, 2019. Sunflower pan-genome analysis shows that hybridization altered gene content and disease resistance. Nature Plants 5, 54–62. https://doi.org/10.1038/s41477-018-0329-0
Layat, E., Leymarie, J., El-Maarouf-Bouteau, H., Caius, J., Langlade, N., Bailly, C., 2014. Translatome profiling in dormant and nondormant sunflower (Helianthus annuus) seeds highlights post - transcriptional regulation of germination. New Phytologist 204, 864–872.
Leroux, D., Rahmani, A., Jasson, S., Ventelon, M., Louis, F., Moreau, L., Mangin, B., 2014. Clusthaplo: a plug-in for MCQTL to enhance QTL detection using ancestral alleles in multi-cross design. Theoretical and applied genetics 127, 921–933.
Louarn, J., Boniface, M.-C., Pouilly, N., Velasco, L., Pérez-Vich, B., Vincourt, P., Muños, S., 2016. Sunflower Resistance to Broomrape (Orobanche cumana) Is Controlled by Specific QTLs for Different Parasitism Stages. Forehead. Plant Sci. 7. https://doi.org/10.3389/fpls.2016.00590
Luoni, SAB, Cenci, A., Moschen, S., Nicosia, S., Radonic, LM, Garcia, JS y, Langlade, NB, Vile, D., Rovere, CV, Fernandez, P., 2021. Genome- Wide Analysis of NAC Transcription Factors in Sunflower (Helianthus Annuus), Their Comparative Phylogenetic Analysis and Association With Leaf Senescence. BMC Plant Biology. https://doi.org/10.21203/rs.3.rs-860249/v1
Mangin, B., Bonnafous, F., Blanchet, N., Boniface, M.-C., Bret-Mestries, E., Carrère, S., Cottret, L., Legrand, L., Marage, G., Pegot-Espagnet, P., Munos, S., Pouilly, N., Vear, F., Vincourt, P., Langlade, NB, 2017a. Genomic Prediction of Sunflower Hybrids Oil Content. Forehead. Plant Sci. 8. https://doi.org/10.3389/fpls.2017.01633
Mangin, B., Casadebaig, P., Cadic, E., Blanchet, N., Boniface, M.-C., Carrère, S., Gouzy, J., Legrand, L., Mayjonade, B., Pouilly, N., André, T., Coque, M., Piquemal, J., Laporte, M., Vincourt, P., Muños, S., Langlade, NB, 2017b. Genetic control of plasticity of oil yield for combined abiotic stresses using a joint approach of crop modeling and genome-wide association. Plant, Cell & Environment 40, 2276–2291. https://doi.org/10.1111/pce.12961
Mangin, B., Casadebaig, P., Cadic, E., Blanchet, N., Boniface, M.-C., Carrère, S., Gouzy, J., Legrand, L., Mayjonade, B., Pouilly, N., André, T., Coque, M., Piquemal, J., Laporte, M., Vincourt, P., Muños, S., Langlade, NB, 2017c. Genetic control of oil yield plasticity to combined abiotic stresses using a joint approach of crop modeling and genome-wide association. Plant, Cell & Environment 40, 2276–2291.
Mangin, B., Pouilly, N., Boniface, M.-C., Langlade, NB, Vincourt, P., Vear, F., Muños, S., 2017d. Molecular diversity of sunflower populations maintained as genetic resources is affected by multiplication processes and breeding for major traits. Theor Appl Genet 130, 1099–1112. https://doi.org/10.1007/s00122-017-2872-x
Mangin, B., Rincent, R., Rabier, C.-E., Moreau, L., Goudemand-Dugue, E., 2019. Training set optimization of genomic prediction by means of EthAcc. PloS one 14, e0205629.
Mangin, B., Sandron, F., Henry, K., Devaux, B., Willems, G., Devaux, P., Goudemand, E., 2015. Breeding patterns and cultivated beets origins by genetic diversity and linkage disequilibrium analyzes . Theor. Appl. Broom. 128, 2255–2271. https://doi.org/10.1007/s00122-015-2582-1
Marchand, G., Huynh‐Thu, VA, Kane, NC, Arribat, S., Varès, D., Rengel, D., Balzergue, S., Rieseberg, LH, Vincourt, P., Geurts, P., Vignes , M., Langlade, NB, 2014. Bridging physiological and evolutionary time-scales in a gene regulatory network. New Phytologist 203, 685–696. https://doi.org/10.1111/nph.12818
Mayjonade, B., Gouzy, J., Donnadieu, C., Pouilly, N., Marande, W., Callot, C., Langlade, N., Muños, S., 2016. Extraction of high-molecular-weight genomics DNA for long-read sequencing of single molecules. BioTechniques 61, 203–205. https://doi.org/10.2144/000114460
Meimoun, P., Mordret, E., Langlade, NB, Balzergue, S., Arribat, S., Bailly, C., El-Maarouf-Bouteau, H., 2014. Is Gene Transcription Involved in Seed Dry After-Ripening ? PLoS ONE 9, e86442. https://doi.org/10.1371/journal.pone.0086442
Moschen, S., Marino, J., Nicosia, S., Higgins, J., Alseekh, S., Astigueta, F., Bengoa Luoni, S., Rivarola, M., Fernie, AR, Blanchet, N., Langlade, NB, Paniego, N., Fernández, P., Heinz, RA, 2019. Exploring gene networks in two sunflower lines with contrasting leaf senescence phenotype using a system biology approach. BMC Plant Biology 19, 446. https://doi.org/10.1186/s12870-019-2021-6
Pecrix, Y., Buendia, L., Penouilh-Suzette, C., Maréchaux, M., Legrand, L., Bouchez, O., Rengel, D., Gouzy, J., Cottret, L., Vear, F ., Godiard, L., 2019. Sunflower resistance to multiple downy mildew pathotypes revealed by recognition of conserved effectors of the oomycete Plasmopara halstedii. Plant J. 97, 730–748. https://doi.org/10.1111/tpj.14157
Penouilh-Suzette, C., Pomiès, L., Duruflé, H., Blanchet, N., Bonnafous, F., Dinis, R., Brouard, C., Gody, L., Grassa, C., Heudelot, X ., Laporte, M., Larroque, M., Marage, G., Mayjonade, B., Mangin, B., Givry, S. de, Langlade, NB, 2020. RNA expression dataset of 384 sunflower hybrids in field condition. OCL 27, 36. https://doi.org/10.1051/ocl/2020027
Rabier, C.-E., Barre, P., Asp, T., Charmet, G., Mangin, B., 2016. On the Accuracy of Genomic Selection. PLOS ONE 11, e0156086. https://doi.org/10.1371/journal.pone.0156086
Rabier, C.-E., Mangin, B., Grusea, S., 2019. On the accuracy in high-dimensional linear models and its application to genomic selection. Scandinavian Journal of Statistics 46, 289–313. https://doi.org/10.1111/sjos.12352
Sahari, K., Nicolas, P., Stéphane, M., Aurélie, B., Fayçal, BJ, Patrick, V., Dominique, B., 2016. Genetic Diversity and Core Collection Constitution for Subsequent Creation of New Sunflower Varieties in Tunisia. Helia. https://doi.org/10.1515/helia-2016-0002
Saux, M., Ponnaiah, M., Langlade, N., Zanchetta, C., Balliau, T., El‐Maarouf‐Bouteau, H., Bailly, C., 2020. A multiscale approach reveals regulatory players of water stress responses in seeds during germination. Plant, Cell & Environment 43, 1300–1313. https://doi.org/10.1111/pce.13731
Terzić, S., Boniface, M.-C., Marek, L., Alvarez, D., Baumann, K., Gavrilova, V., Joita-Pacureanu, M., Sujatha, M., Valkova, D., Velasco, L., Hulke, BS, Jocić, S., Langlade, N., Muños, S., Rieseberg, L., Seiler, G., Vear, F., 2020. Gene banks for wild and cultivated sunflower genetic resources . OCL 27, 9. https://doi.org/10.1051/ocl/2020004
Xia, Q., Saux, M., Ponnaiah, M., Gilard, F., Perreau, F., Huguet, S., Balzergue, S., Langlade, N., Bailly, C., Meimoun, P., Corbineau, F., El-Maarouf-Bouteau, H., 2018. One Way to Achieve Germination: Common Molecular Mechanism Induced by Ethylene and After-Ripening in Sunflower Seeds. International Journal of Molecular Sciences 19, 2464. https://doi.org/10.3390/ijms19082464











